
近日,我校羊遗传改良与生物育种团队王小龙教授联合国内多家单位在国际学术期刊《Nucleic Acids Research》发表了题为“Configuration of adaptable template RNA architectures to unfold the editable space of nuclease prime editor”的研究论文。团队创新性地设计了两种适配核酸酶引导编辑器的新型RNA架构:ActRNA:t双RNA系统和tsp-pegRNA单分子系统,并详细叙述了驱动这些设计的推理过程,突破了现有引导编辑工具的空间限制。
引导编辑技术(PE)作为由CRISPR系统衍生的新一代基因编辑工具,可通过逆转录机制实现精准的基因插入、删除和碱基替换。然而传统PE仅能在DNA切割位点下游的非靶链进行编辑,且编辑效率随距离增加显著降低,限制了其应用潜力。针对这一难题,团队提出了一种全新的“靶向链编辑”策略:设计"靶向链辅助模板RNA"(ActRNA:t),通过sgRNA诱导DNA双链断裂,搭配ActRNA:t实现在切割位点上游的精准编辑。

ActRNA:t和sgRNA协同使用对靶向链进行编辑
项目组通过设计靶向链编程pegRNA(tsp-pegRNA),将引导与编辑功能集成于一个单分子上,该系统在HEK293T细胞中实现17.8-39.7%的精准编辑率,在HeLa、U2OS和HepG2等多种细胞系验证了普适性,并成功模拟了TTR和ASS1基因的致病突变模型。
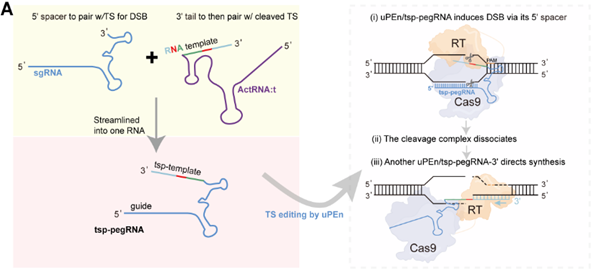
tsp-pegRNA的设计及驱动靶向链编辑的模型
随后,项目组进一步揭示了i53模块通过促进DNA断裂末端的5’端切除机制,可显著提升目标链的可编辑性。联合DNA-PK抑制剂AZD7648后,编辑效率最高可达到75.8%,平均编辑纯度提升了约43倍。经PacBio长读长测序验证,新系统未诱发大规模基因组缺失。
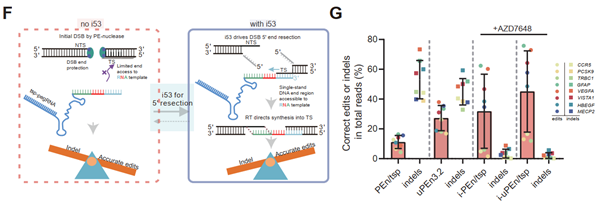
i53在uPEn依赖的TS编辑中的分子机制及AZD7648的协同增效
项目组首次揭示 i53模块的协同机制:通过诱导DNA断裂末端的5'端切除,暴露出靶向链的单链区域,使tsp-pegRNA的PBS序列得以结合并启动逆转录。这一发现颠覆了传统引导编辑器的单复合物作用范式,建立“切割-解离-再编辑”双复合物模型。同时该系统显著拓宽了PE编辑窗口,有效攻克传统难编靶点的效率瓶颈,与传统PE形成正交互补,实现对更广泛人类基因组的高效精准编辑,为基因编辑及动植物生物育种提供了高效的候选工具。
我校博士生陈平博和浙江大学博士后李向阳为论文共同第一作者,我校王小龙教授、南京大学刘江怀教授和浙江大学黄行许教授为论文通讯作者,研究得到了羊遗传改良与生物育种团队负责人陈玉林教授的大力支持。该研究得到国家重点研发计划、国家自然科学基金、国家现代农业产业技术体系、陕西省两链融合重大专项等项目资助。
原文链接:https://academic.oup.com/nar/article/53/11/gkaf522/8160308#
编辑:张晴
终审:刘玉峰